打破领域瓶颈!张锋再发Science
研发家
|
2023-11-24
0
微生物系统是包括CRISPR在内的许多生物技术的基础,但是序列数据库的指数级增长使得很难找到以前未识别的系统。
2023年11月23日,博德研究所张锋及美国国立卫生研究院Eugene V. Koonin共同通讯在Science 在线发表题为“Uncovering the functional diversity of rare CRISPR-Cas systems with deep terascale clustering”的研究论文,该研究开发了基于位置敏感哈希的快速聚类(FLSHclust)算法,该算法在线性时间内对大量数据集进行深度聚类。
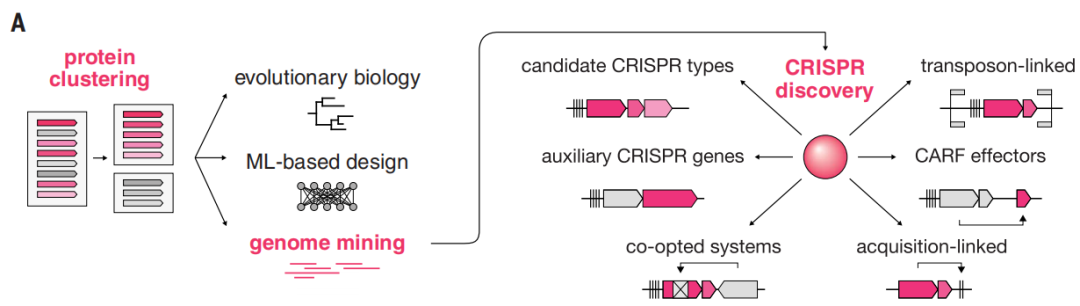
该研究将FLSHclust纳入CRISPR发现管道,并鉴定了188个以前未报道的CRISPR相关基因模块,揭示了许多与适应性免疫相关的其他生化功能。该研究通过实验表征了三种含HNH核酸酶的CRISPR系统,包括第一种具有特定干扰机制的IV型系统,并对它们进行了基因组编辑。该研究还鉴定并表征了一种候选的VII型系统,显示了它对RNA的作用。这项工作为利用CRISPR和更广泛地探索微生物蛋白质的巨大功能多样性开辟了新的途径。
最后,iNature在2023年11月18日总结了张锋在2023年发表的Science,Nature,Cell 等发表的8篇文章。
酶和天然生化系统的发现推进了分子进化研究,揭示了生物过程,为分子技术的发展提供了一个起点。在过去的几十年里,通过对快速增长的核酸和蛋白质序列数据库的系统挖掘,发现了种类繁多的蛋白质家族和功能系统。这些工作中有许多使用蛋白质聚类来对大型数据集中的相似序列进行分组。然后,这些算法的输出可用于蛋白质序列的深度学习、三维(3D)蛋白质结构预测和基因组挖掘。后者的一个主要例子是发现了以前未知的CRISPR系统,它导致了变革性生物技术和治疗方法的发展。
CRISPR系统是微生物RNA引导的适应性免疫系统。它们由CRISPR阵列组成,该阵列编码产生向导的CRISPR RNA (crRNA);一个适应模块,将新的间隔器集成到CRISPR阵列中;以及干扰模块,该干扰模块由由crRNA引导的效应组件组成,该效应组件随后被切割到匹配的靶标。CRISPR效应物可以是1类CRISPR系统中的Cas蛋白复合物(例如Cascade),也可以是2类CRISPR系统中的单、多结构域蛋白(例如Cas9、Cas12或Cas13)。CRISPR系统固有的模块化和可编程性已经被用来开发一套RNA引导的分子技术,从Cas9介导的基因组编辑开始。
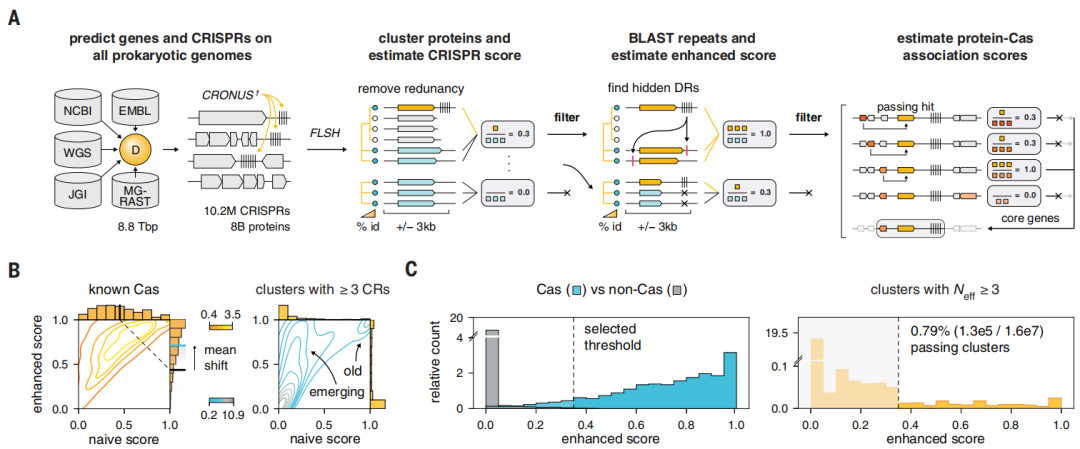
FLSHclust的设计与实现(图源自Science )
这个工具箱已经通过计算搜索得到扩展,发现了许多CRISPR系统。然而,现有的方法依赖于具有二次运行时间的算法,例如对所有比较和蛋白质聚类,这对于挖掘包含数十亿蛋白质的指数增长数据集来说很快变得不切实际。线性缩放聚类方法,如LinClust,可以解决其中的一些问题,但是产生的高度相似序列的小聚类限制了研究深层进化关系的能力。蛋白质结构域谱,如PFAM,可以用来识别广泛丰富的关联,但它们将远程同源物分组,这导致虚假的关联而缺失罕见的关联。
发现数百个罕见的以前未被发现的CRISPR系统,具有敏感的,可扩展的CRISPR关联管道(图源自Science )
为了解决这些限制并利用已知蛋白质结构和功能多样性的爆炸性增长,该研究开发了快速基于位置敏感哈希的聚类(FLSHclust)(发音为“flash clust”),这是一种线性缩放的并行深度聚类算法,O(N logN)。FLSHclust可以处理数十亿个蛋白质,能够对庞大的、指数级增长的序列数据库进行有效分析。该研究应用FLSHclust来鉴定以前未被鉴定的CRISPR系统,包括候选的VII型CRISPR系统,生成RNA引导蛋白目录,扩展我们对这些系统的生物学和进化的理解,并为开发新的生物技术提供起点。
版权及免责声明:本网站所有文章除标明原创外,均来自网络。登载本文的目的为传播行业信息,内容仅供参考,如有侵权请联系删除。文章版权归原作者及原出处所有。本网拥有对此声明的最终解释权